비지도 학습, 데이터 전처리 스터디 내용 요약
업데이트:
3. 비지도학습과 데이터 전처리
비지도 학습
unsupervised learning
비지도 학습의 종류
- 비지도 변환(unsupervised transformation)
- 데이터를 새롭게 표현하여 사람이나 다른 머신러닝 알고리즘이 쉽게 해석할 수 있도록 만드는 알고리즘
- 대표적으로는, 차원 축소(dimensionality reduction)
- 고차원 데이터의 피처 수를 줄이고, 꼭 필요한 데이터만 남기는 방법
- 대표적인 차원 축소로 시각화를 위해 데이터셋을 2차원으로 변경하는것
- 데이터 구성 단위나 성분을 찾기도 함
- ex. 텍스트 문서에서 주제를 추출하는 일
- 군집 알고리즘
- 데이터를 비슷한 것끼리 그룹으로 묶는 것
- ex. sns 사진 분류
- 찍은 사람 뿐만 아니라, 사진의 얼굴이 누구인지 군집화
- 데이터를 비슷한 것끼리 그룹으로 묶는 것
비지도 학습의 도전 과제
- 비지도 학습에서 어려운 점들
- 유용한 것을 학습했는가?
- 이게 올바른 출력인가?
- 그러니까, 모델이 일을 잘 하고있는가?
- ex. 군집 알고리즘이 옆모습, 앞모습을 분류하였음 → 원하는 방향은 아님.
- 그런데 위와 같은 상황에서 우리가 원하는 분류의 기준을 알려줄 수가 없음
- 학습 결과를 평가하기 위해 직접 확인하는 것이 유일한 방법인 경우가 많음
- 비지도 학습 알고리즘은 EDA단계에서 많이 사용
- 지도 학습의 전처리 단계에서도 사용
- 비지도 학습의 결과로 새롭게 표현된 데이터를 사용하여 학습하면
- 정확도가 좋아지는 경우도 있고
- 시간과 메모리 절약 가능
- 비지도 학습의 결과로 새롭게 표현된 데이터를 사용하여 학습하면
데이터 전처리와 스케일 조정
- 간단한 예시 살펴보기
mglearn.plots.plot_scaling()여러 가지 전처리 방법
- StandardScaler
- Z표준화, 평균0, 분산1
- (x - 평균) / 표준편차
- 피처의 최솟값과 최댓값 크기 제한하지 않음
- Z표준화, 평균0, 분산1
- RobustScaler
- 특성들이 같은 스케일 갖게됨
- StandardScaler과 달리, 중간 값(median)과 사분위 값(quartile)을 사용
- (x - q₂(median)) / (q₃ - q₁) (데이터에서 중간값 빼고 q₃ - q₁범위로 나눔)
- 이상치(outlier)에 영향을 받지 않고 로버스트함
- MinMaxScaler
- 모든 데이터가 0과 1 사이에 위치하도록 데이터 변경
- x - x_min / x_max - x_min (데이터에서 최솟값을 빼고 전체 범위로 나눔)
- 모든 데이터가 0과 1 사이에 위치하도록 데이터 변경
- Normalizer
- 피처 벡터의 유클리디안 길이가 1이 되도록 데이터 포인트 조정
- 지름이 1인 원에 데이터들을 투영
- 데이터가 다른 비율로 스케일이 조정됨
- 이런 정규화(Normalization)는 피처 벡터의 길이는 상관 없고 방향(각도)이 중요할 때 사용
- 데이터 변환하기
- 항상 훈련 세트와 테스트 세트에 같은 변환을 적용해야함
- 테스트 세트에 transform 메서드 적용될 때, 테스트 세트의 최소, 최댓값이 아닌 훈련 세트의 그것들에 적용됨
- x_test - x_train_min / x_train_max - x_train_min ```python import sklearn.preprocessing import MinMaxScaler
scaler = MinMaxScaler() # StandardSclaer(), RobustScaler(), Normalizer()
# y_train은 넣지 않음 # 스케일링한 결과를 원하지는 않으니까! scaler.fit(X_train)
# 실제 데이터 변환은 transform 메서드로 X_train_scaled = scaler.transform(X_train) X_test_scaled = scaler.transform(X_test)
# 더 효율적인 방법을 사용해보자 scaler = MinMaxScaler() X_train_scaled = scaler.fit(X_train).transform(X_train) # 방법 1 X_train_scaled = scaler.fit_transform(X_train) # 방법 2, 더 효율적 X_test_scaled = scaler.transform(X_test) ```
차원 축소, 특성 추출, 매니폴드 학습
- 항상 훈련 세트와 테스트 세트에 같은 변환을 적용해야함
- 비지도학습을 사용하는 가장 일반적인 이유
- 시각화
- 데이터 압축
- 추가 처리 (주로 지도 학습에 사용하기 위해서)
주로 학습할 것
- 주성분 분석(PCA, principal component analysis)
- 위와 같은 용도로 가장 간단하고 흔히 사용하는 알고리즘
- 비음수 행렬 분해(NMF, non-negative matrix factorizaion)
- 특성 추출에 널리 사용
- t-SNE(t-distributed stochastic neighbor embedding)
- 2차원 산점도를 이용해 시각화 용도로 많이 사용
주성분 분석(PCA)
- 피처들이 통계적으로 상관관계가 없도록 데이터셋을 회전시키는 기술
-
회전 뒤 데이터를 설명에 있어 중요도에 따라 종종 새로운 특성 중 일부만 선택
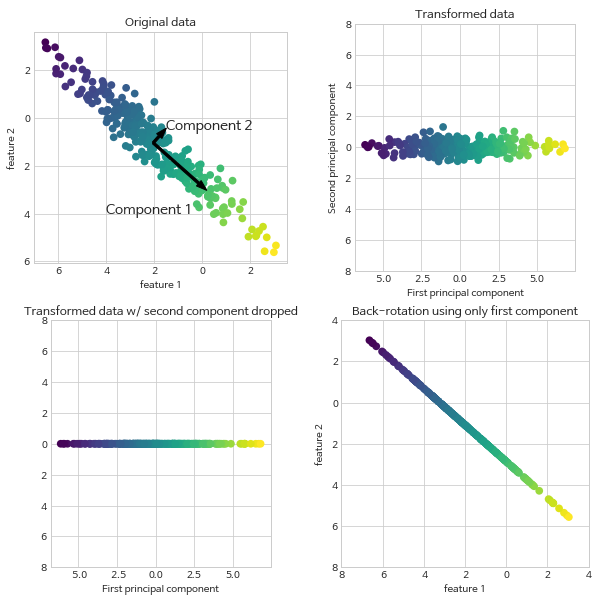
- 분산이 가장 큰 방향을 찾음 (그림(1,1)에서 Component1 방향)
- 상관관계가 큰 방향
- 찾은 방향에서 직각이 되는 방향 중 가장 정보를 많이 포함한 방향 찾음
- 고차원에선 수많은 직각 방향이 존재
- 주성분(principal component)
- 주된 분산의 방향을 주성분이라고 함
- 그림에서 화살표의 방향은 사실 의미 없음. (Com1이 왼쪽 위든, 오른쪽 아래든)
- 일반적으로 원본의 피처의 개수만큼의 주성분 존재
- 데이터에서 평균을 빼고 회전(그림(1,2)), 중심을 원점에 맞춤
- 두 축은 연관되어있지 않음
- 변환된 데이터의 상관관계 행렬(correlation matrix)이 대각선 방향을 제외하고는 0이 됨
- 상관관계 행렬: 공분산 행렬을 정규화한 것
- 주 성분의 일부만 남겨 차원 축소 용도로 사용 가능 (그림(2,1))
- 단순히 일부만 남기는 것이 아님
- 가장 유용한 방향을 찾아, 그 방향의 성분을 유지 (그림에선 첫 번째 성분 유지)
- 데이터에 평균을 더한 후 다시 회전(그림(2,2))
- 이 데이터들은 원래 특성 공간에 놓여있지만, 축소된 차원의 성분만 가지고 있음
- 이 과정에서 노이즈를 제거하거나 주성분에서 유지되는 정보 시각화하는데 종종 쓰임
-
-
가장 널리 사용되는 분야는 고차원 데이터셋 시각화
- PCA를 이용한 데이터 시각화
- 해석하기가 쉽지는 않다는 단점
- PCA를 이용한 시각화는 노트북 참조
from sklearn.decomposition import PCA
# 처음 두 개의 주성분만 유지
pca = PCA(n_components=2)
# PCA모델 생성
pca.fit(X_scaled)
# 처음 두 개의 주성분을 사용해 데이터 변환
X_pca = pca.transform(X_scaled)
# 이후 산점도로 나타내보기
- PCA객체가 학습(fit)될 때 components_속성에 주성분 저장됨
-
주성분 시각화
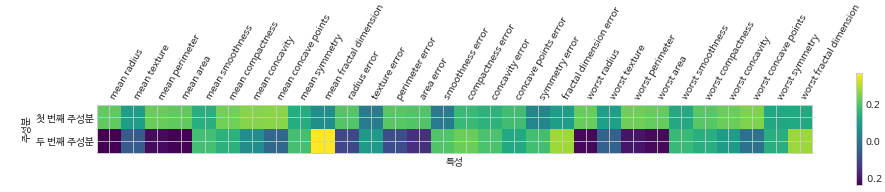
- 첫 번째 주성분은 모두 부호가 같음 (화살표 방향, 즉, 양수냐 음수냐는 의미가 없음)
- 모든 특성 사이에 공통의 송호관계가 있다는 뜻
- 두 번째 주성분은 부호가 섞여있고, 두 주성분 모두 30개의 특성이 있음
- 모든 특성이 섞여있기 때문에, 축이 가지는 의미를 설명하기 쉽지 않다
- 첫 번째 주성분은 모두 부호가 같음 (화살표 방향, 즉, 양수냐 음수냐는 의미가 없음)
- 고유얼굴(eignenface) 특성 추출
- 얼굴인식
- 새로운 얼굴 이미지가 DB에 있는지 찾는 작업
- 해결 방법1
- 각 사람을 다른 클래스로 분류하는 분류기 생성
- 이 경우, 보통 사람 수는 많지만, 1인 당 사진 수는 적음 - 훈련 데이터가 적다
- 그럼, 대규모 모델을 다시 훈련시키지 않고 새로운 얼굴을 쉽게 추가하는 방법 없을까?
- 1-최근법 이웃 분류기
from sklearn.neighbors import KNeighborsClassifier # 데이터를 훈련 세트와 테스트 세트로 나눔 X_train, X_test, y_train, y_test = train_test_split( X_people, y_people, stratify=y_people, random_state=0) # 이웃 개수를 한 개로 하여 KNeighborsClassifier 모델을 만들기 knn = KNeighborsClassifier(n_neighbors=1) knn.fit(X_train, y_train) print("1-최근접 이웃의 테스트 세트 점수: {:.2f}".format(knn.score(X_test, y_test))) # 0.23- 원본 픽셀 공간에서 거리를 계산하는 것은 효과적이지 않음
- 새로운 이미지에서 얼굴이 조금만 오른쪽으로 가도 다른 사진으로 분류하기 때문
- 주성분으로 변환하여 거리를 계산하면 정확도가 높아지지 않을까?
- PCA의 화이트닝 옵션
-
주성분의 스케일이 같아지도록 조정 (변환한 후 StandardScaler를 적용하는 것과 같음)
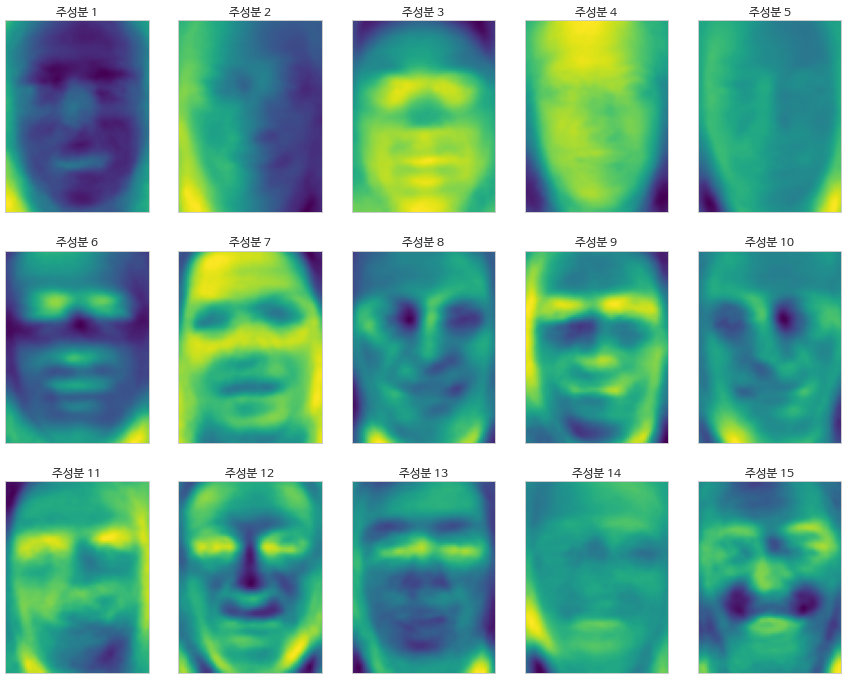
- 사람이 이해하는것과 알고리즘이 데이터를 해석하는 방식은 상당히 다르다.
- 특히 이미지와 같은 경우엔 더더욱!
-
주성분을 이해하기 위해, 몇 개의 주성분을 사용해 원본 데이터를 재구성해보기(무서어)

-
- 얼굴인식
비음수 행렬 분해(NMF, non-negative matrix factorization
- 유용한 특성을 뽑아내기 위한 알고리즘
- PCA와 비슷, 차원 축소에 사용할 수 있음
- 섞여있는 데이터에서 원본 성분을 구분 가능
- 예를들어 여러 사람의 목소리 오디오 데이터 → 목소리 구분
- 여러 악기로 이뤄진 음악
- NMF에서는 음수가 아닌 성분과 계수 값을 찾음
- 주성분과 계수가 모두 0보다 크거나 같아야함
- 음수가 아닌 피처를 가진 데이터에만 적용 가능!
- 데이터가 (0,0)에서 가는 방향!
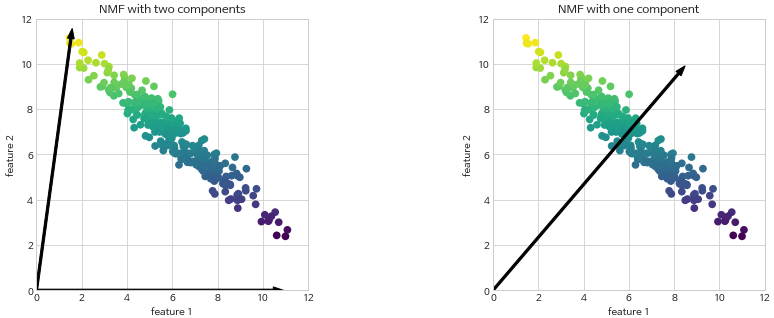
- 하나의 성분을 사용한다면, 데이터를 가장 잘 표현하는 성분으로 향함
- 성분 수를 줄이면 특정 방향이 제거되는 것 뿐만 아니라 전체 성분이 바뀜!
- PCA는 성분이 바뀌지는 않음
- NMF는 성분이 특정 방식으로 정렬도 되어있지 않아, 순서도 없음 (파이썬 set처럼)
t-SNE를 이용한 매니폴드 학습
- 매니폴드 학습 (manifold learning) 시각화 알고리즘들은 복잡한 매핑을 만들어 PCA보다 더 나은 시각화 제공
- 그 중에서도 t-SNE (t-Distributed Stochastic Neighbor Embedding)을 많이 사용
- 목적이 시각화라, 3개 이상의 피처를 뽑는 경우가 거의 없음
- 변환 적용한 훈련 데이터에만 적용 가능, 테스트 세트에는 적용 불가.
- 새로운 세트에 적용 불가
- EDA에는 유용하나, 지도학습용으로는 사용 X
군집(clustering)
- 데이터셋을 클러스터(cluster)라는 그룹으로 나누는 작업
- 그룹내 유사, 그룹간 이질적
- 분류 알고리즙과 비슷하게, 각 데이터 포인트가 어느 클러스터에 속하는지 할당(예측)
k-평균 군집(k-means clustering)
- 데이터의 어떤 영역을 대표하는 영역 찾음
- 데이터 포인트를 가장 가까운 클러스터 중심에 할당
- 클러스터에 할당된 데이터들의 평균으로 중심 재설정
-
2~3 반복 후 클러스터에 할당되는 데이터 포인트에 변화 없을 시 종료
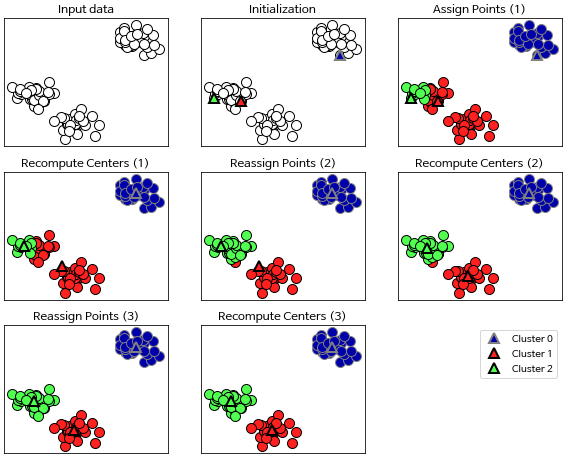
-
새로운 데이터 포인트가 주어지면, 가장 가까운 클러스터 중심을 할당
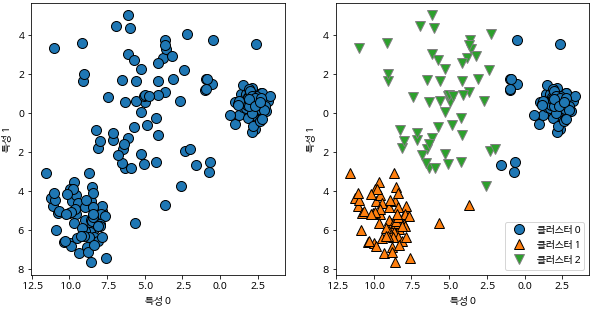
from sklearn.cluster import KMeans kmeans = KMeans(n_cluster=3) # 모델 생성 kmeans.fit(X) # 레이블 확인: labels_ 속성 print('레이블 확인: {}'.format(kmeans.labels_)) # [0, 0, 1, 2, 0, 2, 1, 1, 2 ...] # 클러스터 센터 확인 kmeans.cluster_centers_
-
- 레이블 자체에는 큰 의미가 없고, 의미를 알기 위해선 직접 데이터 확인해봐야함
- 같은 데이터로 다시 훈련시켰을 때, 분류 결과는 같지만 레이블 이름은 바뀔수도 있음
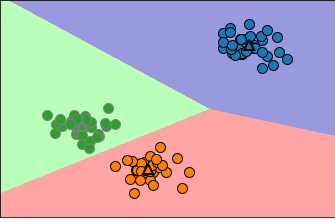
- k-평균 알고리즘이 실패하는 경우
- 데이터셋의 클러스터 수를 알고있더라도 항상 구분할 수 있는 것은 아님
- 클러스터 중심이 하나뿐이므로, 클러스터는 둥근 형태 나타냄
- 그래서 이 알고리즘은 비교적 간단한 형태를 구분 가능
- 또한, k-평균은 모든 클러스터 반경이 똑같다고 가정
-
그 중심 사이의 정확히 중간에 경계 그림
- 클러스터 0, 1을 보면 중심에서 조금 떨어진 데이터도 포함함

- 원형이 아닌 데이터셋에선 잘못 구분할 수 있음
- 모든 방향을 중요시함
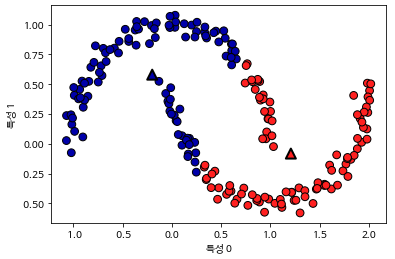
- 복잡한 모양에서도 구분을 잘 하지 못함
- 벡터 양자화(vector quantization)
- PCA: 분산이 큰 방향 찾기
- NMF: 데이터의 극단 또는 일부분에 상응되는 중첩할 수 있는 성분 찾기
- k-means: 클러스터 중심, 즉 데이터들을 하나의 성분으로 표현
- 이렇게 데이터들을 하나의 성분(중심)으로 분해하는 관점으로 보는 것을 벡터 양자화
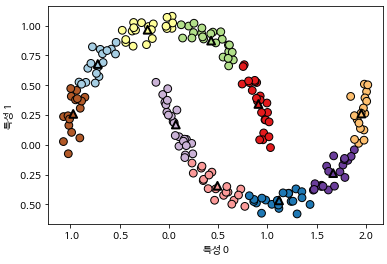
- k-means는 차원보다 높은 클러스터 지정 가능
- transform(X)하여, 데이터와 클러스터 중심간의 거리 표현 가능
distance_features = kmeans.transform(X) print("클러스터 거리 데이터의 형태:", distance_features.shape) print("클러스터 거리:\n", distance_features)
- 단점
- k-means의 난수 초깃값에 따라 출력이 달라짐
- 클러스터의 모양을 가정하고있어, 활용 범위 제한적
- 클러스터의 수를 지정해줘야함.
- 실무에서는 클러스터 수를 모르는 경우가 많을것임
- 이러한 단점을 개선한 알고리즘은 없을까?
병합 군집(agglomerative clustering)
- 각 포인트를 하나의 클러스터로 지정
- 종료 조건을 만족할 때까지 가장 비슷한 두 클러스터를 합침
- sklearn에서 사용하는 종료 조건은 클러스터 수
- linkage(연결) 옵션에서 ‘비슷한’을 어떻게 측정할 지 지정
- ward(default) → 분산을 가장 작게 증가시키는 두 클러스터를 합침 → 그래서 크기가 비교적 비슷한 클러스터가 생성됨
- average → 포인트 사이의 평균 거리가 가장 짧은 두 클러스터를 합침
- complete(최대연결)
**→ 포인트 사이의 최대 거리가 가장 짧은 두 클러스터를 합침
- 각 클러스터에 속한 포인트 수가 많이 다를 때에 2, 3번의 방법이 나을 수 있음
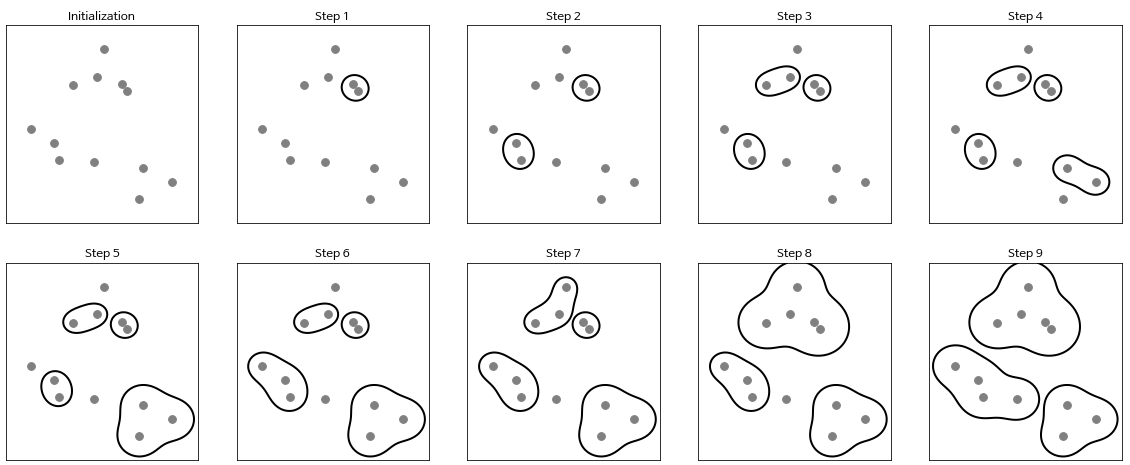
- 새로운 데이터 포인트에 대해서는 예측을 할 수 없음
- predict 메서드 없음
- 대신 훈련 세트로 모델 생성 후, 클러스터 소속 정보를 얻기 위해 fit_predict 메서드 사용
- 여전히 클러스터 수를 지정해줘야하는 문제, 해결할 수 없을까?
계층적 군집과 덴드로그램
-
병합 군집은 계층적 군집을 만듦(hierarchical clustering)

- 덴드로그램(dendrogram)
- 다차원의 계층 군집을 시각화
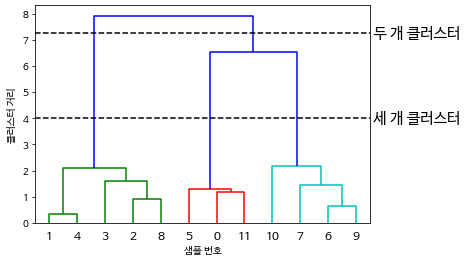
- 가지의 길이: 클러스터 간 거리
- 트리모델과 비슷하게 leaf들과 노드같은 것들로 묶여있음
- 하지만 여전히 복잡한 데이터셋은 구분하지 못함
- 복잡한 것들도 구분해줄 수 있는 알고리즘 없나?
DBSCAN(density-based spatial clustering of applications with noise)
- 장점
- 클러스터 개수를 미리 지정하지 않아도 됨
- 복잡한 세트에서도 구분 가능
- 클래스에 속하지 않는 데이터 포인트 구분 가능
- 병합 군집이나 k-means 보다는 느리지만, 큰 데이터에서도 가능
- 밀집 지역(dense region)
- DBSCAN에서는 데이터가 밀집된 지역을 찾음
- 데이터의 밀집 지역이 한 클러스터를 구성
- 비교적 비어있는 지역을 경계로 다른 클러스터와 구분
- 핵심 샘플(핵심 포인트)
- 밀집 지역에 있는 데이터 포인트
- 매개변수
- min_samples, eps
- 한 데이터 포인트에서 eps 거리 안에 데이터가 min_samples 만큼 들어있으면 그 데이터 포인트를 핵심 샘플로 분류
- eps보다 가까운 핵심 샘플은 DBSCAN에 의해 동일한 클러스터로 합쳐짐
- 작동 순서
- 시작할 때 무작위로 포인트 선택
- eps 거리 안의 모든 포인트 찾음
- eps 거리 안의 포인트 수가 min_samples보다 적으면 잡음(noise)으로 분류
- min_samples보다 많으면, 핵심 포인트로 레이블, 새로운 클러스터 레이블 할당
- 레이블 할당된 데이터들로부터 eps거리 안의 모든 이웃 살핌
- 어떤 레이블에도 할당이 안되어있으면, 전에 만든 레이블 할당
- 만약 핵심 샘플이면, 그 포인트의 이웃을 차례로 방문
- eps 거리 안에 핵심 샘플이 없을때까지 진행
- DBSCAN에서 포인트 종류
- 핵심 포인트
- 경계 포인트(핵심 포인트에서 eps 거리 안에 있는 포인트)
- 잡음 포인트(noise)
- DBSCAN을 한 데이터셋에 여러번 적용하면?
- 핵심 포인트, 잡음 포인트는 동일
- 경계 포인트는 방문 순서에 따라 클러스터 레이블이 다를 수 있음
- 하지만 중요한 이슈는 아니고, 경계 포인트 수 자체가 많지도 않음
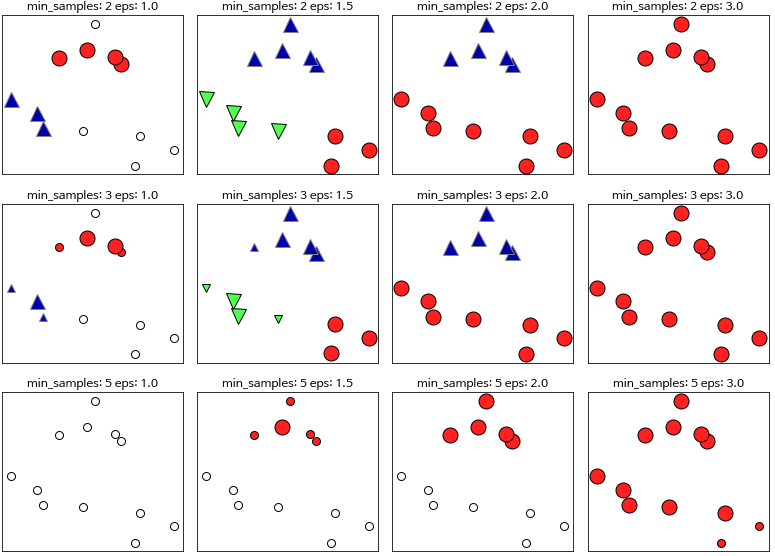
- eps
- 가까운 포인트의 범위를 결정
- 너무 크면 하나의 클러스터에 속하고, 너무 작으면 핵심포인트를 만들지 못하거나 모두 잡음
- 적절값을 잘 찾기 위해선 MinMaxScaler나 StandardScaler로 조정
- min_samples
- 데이터가 덜 모인 지역의 포인트를 잡음처리할 것인지, 클러스터로 속하게 할 것인지 결정
- 레이블 할당값을 조심해야한다!
- 클러스터 레이블을 그대로 사용할 경우, 노이즈인 -1값이 어떤 결과를 불러올지 모름
군집 알고리즘 비교 및 평가
군집 알고리즘은 알고리즘이 잘 작동하는지 평가하거나 비교하기가 매우 어렵다.
- 타깃값으로 군집 평가하기
- 군집 알고리즘의 결과를 실제 정답 클러스터와 비교
- 1(최적)과 0(무작위 분류)사이의 값을 제공하는
- ARI(adjusted rand index) > 값이 음수가 될 수도 있음
- NMI(normalized mutual information)
- 가장 많이 하는 실수는
- adjusted_rand_score나 normalized_mutual_info_score와 같은 군집용 측정 도구를 사용하지 않고, accuracy_score를 적용하는 것
- 정확도(accuracy_score)는 클러스터의 레이블 이름이 실제 레이블 이름과 맞는지 확인
- 클러스터 레이블은 이름에 의미가 없기때문에 결과가 다를 수 있음
from sklearn.metrics import accuracy_score # 포인트가 클러스터로 나뉜 두 가지 경우 clusters1 = [0, 0, 1, 1, 0] clusters2 = [1, 1, 0, 0, 1] # 모든 레이블이 달라졌으므로 정확도는 0 print("정확도: {:.2f}".format(accuracy_score(clusters1, clusters2))) # 같은 포인트가 클러스터에 모였으므로 ARI는 1 print("ARI: {:.2f}".format(adjusted_rand_score(clusters1, clusters2))) # 정확도: 0.00 # ARI: 1.00
- 클러스터 레이블은 이름에 의미가 없기때문에 결과가 다를 수 있음
- 실루엣 계수(silhouette coefficient)
- 근데 사실 타깃값이 없는 경우가 더 많음 (알면 바로 분류기 모델을 사용하면 되는디)
- 클러스터의 밀집 정도를 계산하는 것
- 높을수록 좋으며 최대는 1
- 그런데 이 계수도 잘 동작하지는 않으며, 복잡한 경우에는 평가가 잘 맞지 않음
- 가장 좋은 평가 전략은 견고성 기반 지표 (robustness-based)
- sklearn에는 아직 없음
- 데이터에 노이즈를 추가하거나 여러가지 매개변수 설정으로 알고리즘 실행, 결과 비교
- 매개변수등을 변화시켜 반복했을 때 결과가 일정하면 신뢰 가능
- 결국 클러스터가 내가 원하는대로 구분했는지 보려면 직접 확인해야함
- 다른 방법 찾아보기
- 이상치 검출(outlier detection)
- 특이한 것을 찾아내는 것
- 군집화 중 노이즈에 해당되는 데이터들을 확인해보니 이상한 사진들인 경우 등..
- 군집 알고리즘 요약
- 종류
- k-means, DBSCAN, 병합 군집
- k-means, 병합 군집은 클러스터 수 지정 가능
- DBSCAN은 eps와 클러스터 크기 매개변수로 조정 가능
- k-means
- 클러스터 중심을 사용해 구분
- 클러스터 중심이 각 클러스터 데이터들을 대표하기 때문에, 분해로도 볼 수 있음
- DBSCAN
- 클러스터에 할당되지 않는 잡음 포인트 인식 가능
- 클러스터 수 자동 결정
- 복잡한 클러스터의 모양 인식 가능(two_moons예시)
- 크기가 많이 다른 클러스터를 만들어내곤 함(장,단점 공존)
- 병합 군집
- 전체 데이터의 분할 계층도 생성
- 덴드로그램으로 시각화 가능
- 종류
댓글남기기